دوره 34، شماره 4 - ( 4-1402 )
جلد 34 شماره 4 صفحات 234-223 |
برگشت به فهرست نسخه ها
Ethics code: IR.PNU.REC.1401.482
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Gholamalaian D, Zare M. ANALYSIS OF CLOCK GENE RS1801260 POLYMORPHISM IN IRANIAN PATIENTS WITH INSOMNIA. Studies in Medical Sciences 2023; 34 (4) :223-234
URL: http://umj.umsu.ac.ir/article-1-6002-fa.html
URL: http://umj.umsu.ac.ir/article-1-6002-fa.html
غلامعلیان دلارام، زارع مریم. بررسی پلیمورفیسم rs1801260 ژن CLOCK در بیماران ایرانی مبتلا به بیخوابی (اینسومنیا). مجله مطالعات علوم پزشکی. 1402; 34 (4) :223-234
استادیار، گروه زیست شناسی، دانشکده علوم پایه، دانشگاه پیام نور، تهران، ایران (نویسنده مسئول) ، mariamzare@yahoo.com
متن کامل [PDF 573 kb]
(854 دریافت)
| چکیده (HTML) (3269 مشاهده)
یافتهها
.jpg)
.jpg)
در این مطالعه 120 نمونه خون (60 بیمار و 60 کنترل نرمال) موردبررسی قرارگرفت. فراوانی و درصد پلیمورفیسم rs1801260 در ژن CLOCK در دو گروه بیمار و کنترل در جدول 2 نشان داده شده است. میزان بروز ژنوتیپ هموزیگوت موتانت GG در افراد بیمار 50درصد و در افراد سالم 30درصد است. میزان بروز ژنوتیپ هتروزیگوت AG در افراد بیمار 7/36درصد و در افراد سالم 7/26درصد است. درحالیکه میزان بروز ژنوتیپ هموزیگوت نرمال AA در افراد بیمار تنها 3/13درصد بوده که این میزان در افراد نرمال به 3/43درصد میرسد (جدول 2)(شکل 4).
جدول (3): بررسی رابطه بین پلیمورفیسم rs1801260 و خطر بروز بیماری در دو گروه بیمار و کنترل
ارتباط آماری بین جنسیت بیماران و پلیمورفیسم rs1801260 نیز موردبررسی قرار گرفت. همانطور که در جدول 4 نشان داده شده است، ازآنجاکه سطح معنادار آماری (P value) بیشتر از 0/05 در نظر گرفتهشده، بنابراین بین پلیمورفیسم rs1801260 و جنسیت بیماران ارتباط معنیداری وجود ندارد (P=0.924). بهعبارتدیگر پلیمورفیسم rs1801260 بیماران مستقل از جنسیت است. همچنین ارتباط بین سبک زندگی افراد بیماران و وجود پلیمورفیسم rs1801260 ژن CLOCK نیز موردبررسی قرار گرفته است. نتایج حاصله نشان داده است که بین پلیمورفیسم rs1801260 و سبک زندگی بیماران ارتباط معنیداری وجود ندارد (P=0.270). بهعبارتدیگر پلیمورفیسم rs1801260 در بیماران مستقل از سبک زندگی بیماران است (جدول 4). بررسی آماری ارتباط بین سن افراد موردمطالعه و پلیمورفیسم rs1801260 ژن CLOCK نیز نشان داده است که پلیمورفیسم rs1801260 ارتباط معنیداری با سن بیماران ندارد (P=0.943).
جدول (4): بررسی رابطه بین پلیمورفیسم rs1801260 با جنسیت و سبک زندگی بیماران
بحث و نتیجهگیری
متن کامل: (1492 مشاهده)
مقدمه
ریتمهای شبانهروزی، چرخههای ویژه خوابوبیداری هستند که توسط فرایندهای درونی و فاکتورهای بیرونی تنظیم میشوند. این ریتمهای متناوب 24 ساعته توسط ساعتهای مولکولی ایجاد میشوند و وظیفه هماهنگ کردن زمان درونی بدن با دنیای بیرونی را به عهده دارند. تقریباً تمام فیزیولوژی بدن توسط ریتم شبانهروزی تنظیم میشود و اختلال در آن عواقب عمدهای بر سلامتی دارد (1). در این رابطه، فرایند خواب بهشدت توسط ریتمهای شبانهروزی تنظیم میشود، بهطوریکه ناهماهنگی بین ریتمهای شبانهروزی و محیط خارجی ممکن است منجر به اختلالات خواب ریتم شبانهروزی شود (2). بیخوابی (Insomnia) بهعنوان یکی از مهمترین انواع اختلالات خواب هست که در سراسر جهان بین 16/8 تا 31 درصد از جمعیت عمومی را تحت تأثیر قرار میدهد. بیخوابی با دشواری مداوم در شروع یا حفظ خواب و یا بیداری در صبح زود، همراه با اختلالات عملکردی مرتبط مانند خستگی یا مشکل در تمرکز در طول روز مشخص میشود. خطرات سلامتی مرتبط با بیخوابی شامل اختلالات مزمن و روانی متعددی ازجمله بیماری قلبی، سندرم متابولیک، چاقی، افسردگی و اضطراب است (3، 4). خواب مناسب برای حفظ سلامتی و بهینه سازی عملکرد کلی در هنگام بیداری ضروری است. زمان، عمق و مدت خواب توسط سیستم شبانهروزی و هومئوستاز خواب تنظیم میشود که به مدل دو فرآیندی معروف است (1، 5).
شناسایی تأثیر متقابل ژنتیک بر سلامت خواب و خواب بر بیان و کارکرد صحیح ژنها، برای فهم ریتمهای شبانهروزی و لایههای زیرین آن لازم و ضروری است. ریتم شبانهروزی به تدریج از ماه سوم حیات به بعد با پیدا کردن توانایی تولید ملاتونین در بدن نوزاد تعریف میشود و الگوهای بیولوژیکی بدن ازجمله چرخههای خوابوبیداری، دما، فشار خون، ترشح هورمونها، دفع و غیره را کنترل میکند. این وقایع شبانهروزی توسط ساعتهای بیولوژیکی کنترل میشوند و با هر دو نشانه محیطی مانند نور و دما و همچنین با نشانههای اجتماعی مانند فعالیت بدنی و رفتار تغذیهای هماهنگ میشوند. چرخدندههای این ساعت شبانهروزی پروتئینهایی هستند که تولید و تخریب آنها، توسط مسیرهای فیدبکی به هم پیوسته کنترل میشود (5 و 6). از دیدگاه مولکولی، ساعت شبانهروزی در پستانداران از تعدادی ژن تشکیل شده است که بهصورت لوپ های فیدبکی رونویسی و ترجمهای با هم کار کرده و نوسانات شبانهروزی را تولید میکنند. در مرکز این شبکه مولکولی دو عامل رونویسی وجود دارد: فاکتور (CLOCK) و فاکتور (BMAL1) که با هم هترودایمر میشوند و به پروموترهای ژنهای ساعت و همچنین تعداد زیادی از ژنهای کنترل شده با ساعت متصل شده و باعث بیان ژنهای Period (Per1-3) و Cryptochrome (Cry1/2) میشوند که آنها نیز به نوبه خود با فسفریله شدن و تشکیل کمپلکس و انتقال به هسته بهصورت منفی از فعالیت رونویسی CLOCK-BMAL1 جلوگیری میکنند. این لوپ فیدبک منفی تقریباً 24 ساعت طول میکشد تا تکمیل شود (7).
طبق مطالعات صورت گرفته تغییرات ژنتیکی در این ژنهای ساعت با اثرات فنوتیپی درمجموعهای از فرآیندهای بیولوژیکی مانند ترجیح خواب روزانه، خواب شبانه، متابولیسم، تنظیم خلق و خو، اعتیاد و باروری همراه است. تخمین زده شده است که حداقل 20درصد از کل ژنهای پستانداران با ساعت کنترل میشوند که همین امر نشاندهنده تنظیم گسترده ژن شبانهروزی است (8 و 9). بر اساس مطالعات، خواب ناکافی باعث افزایش بیان ژنهای مرتبط با استرس اکسیداتیو و همینطور پاسخ ایمنی میشود. علاوه بر این، کمبود خواب جزئی با پاسخهای آسیب DNA و پیری سلول و مشکلات حافظه مرتبط است (10، 11).
تاکنون 13 ژن شبانهروزی هستهای که به آنها ژن ساعت (Circadian Clock Genes) نیز اطلاق میشود در انسان شناسایی شده است و در سالهای اخیر، عملکرد این ژنها بهطور قابلتوجهی موردبررسی قرار گرفته است. ژنهای اصلی ساعت در جزایر پانکراس انسان، جایی که انسولین بهطور ریتمیک ترشح میشود، بیان میشوند و این تنظیم به هموستازی گلوکز کمک میکند. (9، 12، 13).
ژن CLOCK (The Circadian Locomotor Output Cycle Kaput) یکی از اولین ژنهایی است که در این رابطه موردبررسی قرار گرفته است. این ژن بر روی بازوی بلند کروموزوم 4q12 قرار دارد و فاکتور رونویسی CLOCK را کد میکند که نقش مرکزی در ساعت شبانهروزی داشته و باعث تنظیم رونویسی بسیاری از ژنهای خروجی ریتم شبانهروزی میشود. اختلال در عملکرد این ژن، تنظیم رونویسی این ژنها را برهم می زند. مطالعات نشان دادهاند که وقوع پلیمورفیسمهای ژنتیکی در عملکرد این ژن تأثیر گذار است (12، 14).
یکی از انواع پلیمورفیسمهای شناسایی شده در این ژن که با فنوتیپ های انسانی مرتبط هستند، پلیمورفیسم rs1801260 A/G در ناحیه ترجمه نشدنی سر ′3 (3’ UTR) ژن CLOCK میباشد. مطالعات نشان دادهاند که پلیمورفیسم این جایگاه با میزان بیان پروتئین CLOCK مرتبط است (15). علاوه بر نقش محوری که ژن CLOCK در ساعت شبانهروزی ایفا میکند، این ژن در تنظیم خواب نیز مشارکت به سزایی دارد. بر اساس برخی شواهد افراد حامل آلل G در این جایگاه، اغلب حدود 10 تا 44 دقیقه تأخیر در زمانبندی ترجیحی خود برای ورود به مراحل فعال خواب دارند و افراد با ژنوتیپ هموزیگوت GG به طرز چشمگیری با کاهش خواب شب و افزایش خوابآلودگی در روز همراه هستند (12، 16).
لازم به ذکر است که ارتباط بین پلیمورفیسم rs1801260 و اختلالات خواب در بیماران مبتلا به اختلالات روانپزشکی نیز مشاهده شده است. در میان بیماران مبتلا به اختلال افسردگی اساسی (Major Depressive Disorde: MDD) یا اختلال دو قطبی (Bipolar Disorder) افراد هموزیگوت حاوی آلل G، با کاهش خواب اساسی و افزایش اختلال خواب مواجهند. بعلاوه مشاهده شده است که ایجاد جهش مداوم در ژن CLOCK در موشها، چهارچوب هموستاز خواب در آنها را تغییر میدهد (17، 18). همچنین شواهد نشان دادهاند که پلیمورفیسمهای ژن CLOCK ازجمله جایگاه rs1801260 با خلق و خو، بیماریهایی مانند بیش فعالی، فرایندهای متابولیکی، چاقی و سرطان نیز مرتبط میباشد (1، 6، 17). لذا با توجه به اهمیت این ژن و نقش مرکزی آن در تنظیم بیان سایر ژنهای ساعت در انسان و نیز با توجه تنوع ژنتیکی این ژن در جوامع مختلف، در این مطالعه پلیمورفیسم جایگاه rs1801260 ژن CLOCK در بیماران مبتلا به اختلال بیخوابی در جمعیت ایرانی موردبررسی قرار گرفت.
شناسایی تأثیر متقابل ژنتیک بر سلامت خواب و خواب بر بیان و کارکرد صحیح ژنها، برای فهم ریتمهای شبانهروزی و لایههای زیرین آن لازم و ضروری است. ریتم شبانهروزی به تدریج از ماه سوم حیات به بعد با پیدا کردن توانایی تولید ملاتونین در بدن نوزاد تعریف میشود و الگوهای بیولوژیکی بدن ازجمله چرخههای خوابوبیداری، دما، فشار خون، ترشح هورمونها، دفع و غیره را کنترل میکند. این وقایع شبانهروزی توسط ساعتهای بیولوژیکی کنترل میشوند و با هر دو نشانه محیطی مانند نور و دما و همچنین با نشانههای اجتماعی مانند فعالیت بدنی و رفتار تغذیهای هماهنگ میشوند. چرخدندههای این ساعت شبانهروزی پروتئینهایی هستند که تولید و تخریب آنها، توسط مسیرهای فیدبکی به هم پیوسته کنترل میشود (5 و 6). از دیدگاه مولکولی، ساعت شبانهروزی در پستانداران از تعدادی ژن تشکیل شده است که بهصورت لوپ های فیدبکی رونویسی و ترجمهای با هم کار کرده و نوسانات شبانهروزی را تولید میکنند. در مرکز این شبکه مولکولی دو عامل رونویسی وجود دارد: فاکتور (CLOCK) و فاکتور (BMAL1) که با هم هترودایمر میشوند و به پروموترهای ژنهای ساعت و همچنین تعداد زیادی از ژنهای کنترل شده با ساعت متصل شده و باعث بیان ژنهای Period (Per1-3) و Cryptochrome (Cry1/2) میشوند که آنها نیز به نوبه خود با فسفریله شدن و تشکیل کمپلکس و انتقال به هسته بهصورت منفی از فعالیت رونویسی CLOCK-BMAL1 جلوگیری میکنند. این لوپ فیدبک منفی تقریباً 24 ساعت طول میکشد تا تکمیل شود (7).
طبق مطالعات صورت گرفته تغییرات ژنتیکی در این ژنهای ساعت با اثرات فنوتیپی درمجموعهای از فرآیندهای بیولوژیکی مانند ترجیح خواب روزانه، خواب شبانه، متابولیسم، تنظیم خلق و خو، اعتیاد و باروری همراه است. تخمین زده شده است که حداقل 20درصد از کل ژنهای پستانداران با ساعت کنترل میشوند که همین امر نشاندهنده تنظیم گسترده ژن شبانهروزی است (8 و 9). بر اساس مطالعات، خواب ناکافی باعث افزایش بیان ژنهای مرتبط با استرس اکسیداتیو و همینطور پاسخ ایمنی میشود. علاوه بر این، کمبود خواب جزئی با پاسخهای آسیب DNA و پیری سلول و مشکلات حافظه مرتبط است (10، 11).
تاکنون 13 ژن شبانهروزی هستهای که به آنها ژن ساعت (Circadian Clock Genes) نیز اطلاق میشود در انسان شناسایی شده است و در سالهای اخیر، عملکرد این ژنها بهطور قابلتوجهی موردبررسی قرار گرفته است. ژنهای اصلی ساعت در جزایر پانکراس انسان، جایی که انسولین بهطور ریتمیک ترشح میشود، بیان میشوند و این تنظیم به هموستازی گلوکز کمک میکند. (9، 12، 13).
ژن CLOCK (The Circadian Locomotor Output Cycle Kaput) یکی از اولین ژنهایی است که در این رابطه موردبررسی قرار گرفته است. این ژن بر روی بازوی بلند کروموزوم 4q12 قرار دارد و فاکتور رونویسی CLOCK را کد میکند که نقش مرکزی در ساعت شبانهروزی داشته و باعث تنظیم رونویسی بسیاری از ژنهای خروجی ریتم شبانهروزی میشود. اختلال در عملکرد این ژن، تنظیم رونویسی این ژنها را برهم می زند. مطالعات نشان دادهاند که وقوع پلیمورفیسمهای ژنتیکی در عملکرد این ژن تأثیر گذار است (12، 14).
یکی از انواع پلیمورفیسمهای شناسایی شده در این ژن که با فنوتیپ های انسانی مرتبط هستند، پلیمورفیسم rs1801260 A/G در ناحیه ترجمه نشدنی سر ′3 (3’ UTR) ژن CLOCK میباشد. مطالعات نشان دادهاند که پلیمورفیسم این جایگاه با میزان بیان پروتئین CLOCK مرتبط است (15). علاوه بر نقش محوری که ژن CLOCK در ساعت شبانهروزی ایفا میکند، این ژن در تنظیم خواب نیز مشارکت به سزایی دارد. بر اساس برخی شواهد افراد حامل آلل G در این جایگاه، اغلب حدود 10 تا 44 دقیقه تأخیر در زمانبندی ترجیحی خود برای ورود به مراحل فعال خواب دارند و افراد با ژنوتیپ هموزیگوت GG به طرز چشمگیری با کاهش خواب شب و افزایش خوابآلودگی در روز همراه هستند (12، 16).
لازم به ذکر است که ارتباط بین پلیمورفیسم rs1801260 و اختلالات خواب در بیماران مبتلا به اختلالات روانپزشکی نیز مشاهده شده است. در میان بیماران مبتلا به اختلال افسردگی اساسی (Major Depressive Disorde: MDD) یا اختلال دو قطبی (Bipolar Disorder) افراد هموزیگوت حاوی آلل G، با کاهش خواب اساسی و افزایش اختلال خواب مواجهند. بعلاوه مشاهده شده است که ایجاد جهش مداوم در ژن CLOCK در موشها، چهارچوب هموستاز خواب در آنها را تغییر میدهد (17، 18). همچنین شواهد نشان دادهاند که پلیمورفیسمهای ژن CLOCK ازجمله جایگاه rs1801260 با خلق و خو، بیماریهایی مانند بیش فعالی، فرایندهای متابولیکی، چاقی و سرطان نیز مرتبط میباشد (1، 6، 17). لذا با توجه به اهمیت این ژن و نقش مرکزی آن در تنظیم بیان سایر ژنهای ساعت در انسان و نیز با توجه تنوع ژنتیکی این ژن در جوامع مختلف، در این مطالعه پلیمورفیسم جایگاه rs1801260 ژن CLOCK در بیماران مبتلا به اختلال بیخوابی در جمعیت ایرانی موردبررسی قرار گرفت.
مواد و روشها
افراد مورد مطالعه شامل 60 نمونه از افراد با مشکلات عمقی بیخوابی، ناتوانی در کنترل خواب، خواب نامنظم و عوارض ناشی از آن در طول روز بهعنوان گروه بیمار و 60 نمونه از افرادی بدون هیچگونه سابقه بیخوابی و یا اختلال در خواب بهعنوان گروه کنترل انتخاب شدند. نمونههای بیمار از افراد مراجعهکننده به کلینیک خواب عرفان تهران طی یک دوره 3 ماهه جمعآوری شد. برای انجام این پژوهش از هر فرد به میزان 5 میلی لیتر خون در لولههای حاوی EDTA استریل گرفته شد و درون ظرف یخ نگهداری و در شرایط استاندارد به آزمایشگاه منتقل شدند. اهداف پژوهش برای همه افراد شرکت کننده توضیح داده شد و تمام افراد بهصورت آگاهانه و با تکمیل و امضا فرم رضایتنامه آگاهانه جهت شرکت در پروژه تحقیقاتی به همراه تأیید اطلاعات آسیبدیدگی توسط متخصص اعصاب و روان در این پژوهش مشارکت نمودند افرادی با سابقه ابتلا به اختلالات و بیماریهای اعصاب و روان و نیز افرادی که سابقه مصرف داروهایی با عوارض بیخوابی داشتهاند، از مطالعه خارج شدند. اطلاعات کلینیکوپاتولوژی بیماران شامل سن، جنس، قد، وزن، BMI و سابقه خانوادگی نیز دریافت شد. تحقیق حاضر توسط کمیته اخلاق دانشگاه پیام نور مورد تأیید قرار گرفته و کد اخلاق پزشکی (IR.PNU.REC.1401.482) دریافت نموده است.
پس از جمعآوری نمونهها، استخراج DNA از خون با استفاده از کیت تکاپوزیست مدل DYnaBio Blood/Tissue DNA Extraction Mini Kit (شرکت تکاپو زیست، ایران) و بر اساس پروتکل مربوطه انجام شد. غلظت و خلوص نمونه DNA استخراج شده، با استفاده از روش کمی طیفسنجی جذبی اشعه ماوراءبنفش و با دستگاه نانودراپ (Nano Drop ND-1000, Thermo Scientific, USA) و نیز الکتروفورز DNA روی ژل 1درصد بررسی شد. سپس برای بررسی وضعیت پلیمورفیسم rs1801260 در ژن CLOCK، تکثیر DNA های استخراج شده با روش ARMS-PCR انجام شد.
برای طراحی پرایمر ابتدا توالی جایگاه rs1801260 با استفاده از پایگاه SNP متعلق به NCBI به دست آمد و سپس طراحی پرایمرهای F و R بهصورت دستی و با کمک سایت NEB (New England Biolabs) انجام شد. توالی پرایمرهای مورد استفاده (شرکت تکاپو زیست، ایران) در جدول 1 نشان داده شده است.
پس از جمعآوری نمونهها، استخراج DNA از خون با استفاده از کیت تکاپوزیست مدل DYnaBio Blood/Tissue DNA Extraction Mini Kit (شرکت تکاپو زیست، ایران) و بر اساس پروتکل مربوطه انجام شد. غلظت و خلوص نمونه DNA استخراج شده، با استفاده از روش کمی طیفسنجی جذبی اشعه ماوراءبنفش و با دستگاه نانودراپ (Nano Drop ND-1000, Thermo Scientific, USA) و نیز الکتروفورز DNA روی ژل 1درصد بررسی شد. سپس برای بررسی وضعیت پلیمورفیسم rs1801260 در ژن CLOCK، تکثیر DNA های استخراج شده با روش ARMS-PCR انجام شد.
برای طراحی پرایمر ابتدا توالی جایگاه rs1801260 با استفاده از پایگاه SNP متعلق به NCBI به دست آمد و سپس طراحی پرایمرهای F و R بهصورت دستی و با کمک سایت NEB (New England Biolabs) انجام شد. توالی پرایمرهای مورد استفاده (شرکت تکاپو زیست، ایران) در جدول 1 نشان داده شده است.
جدول (1): توالی پرایمرهای مورد استفاده برای بررسی پلیمورفیسم rs1801260 ژن CLOCK
| پرایمر | توالی |
| پرایمر Forward (A) | CTAAAACACTGTCAGAACTGGCTA |
| پرایمر Forward (G) | CTAAAACACTGTCAGAACTGGCTG |
| پرایمر Reverse | TGAGATGCAGTATTGAGTGTTC |
برای انجام واکنش ARMS-PCR، برای هریک از نمونههای DNA استخراج شده، دو میکروتیوپ، یکی برای حالت نرمال و دیگری برای حالت موتانت در نظر گرفته شده و واکنش ARMS-PCR با حجم نهایی 20 میکرولیتر و در دستگاه (USA، BioRad) Thermal cycler انجام شد. مخلوط واکنش PCR در دو میکروتیوب جداگانه شامل 20 میکرولیتر از Master mix 2X، 4 میکرولیتر از DNA الگو، 1 میکرولیتر از هرکدام از پرایمرها و 4 میکرولیتر آب عاری از نوکلئاز (سیناکلون، ایران) آماده سازی شد. متعاقباً مخلوط واکنش در دستگاه ترموسایکلر قرار گرفته و واکنش PCR تحت شرایط اختصاصی انجام شد. شرایط و پروفایل دمایی واکنش ARMS-PCR به این صورت بوده است: 1 سیکل برای واسرشتگی اولیه به مدت 5 دقیقه در دمای ºC 95، سپس 34 سیکل تکثیر شامل 30 ثانیه در دمای ºC 95، 30 ثانیه در دمای ºC 62 و 30 ثانیه در دمای ºC 72 و 1 سیکل نهایی در دمای ºC 72 به مدت 5 دقیقه. متعاقباً صحت PCR با انجام الکتروفورز محصولات PCR بر روی ژل آگارز 1 درصد و پس از رنگ آمیزی با اتیدیوم بروماید زیر نور UV تأیید شد. برای تأیید اختصاصی بودن پرایمرها و نتایج به دست آمده، تعیین توالی محصولات PCR نیز انجام شده (شرکت ژنوسل، تهران، ایران) و نتایج بررسی گردید.
نتایج به دست آمده با استفاده از نرمافزار SPSS (version 26) مورد تجزیه و تحلیل آماری قرارگرفت. پس از تعیین فراوانی ژنو تیپهای هموزیگوت نرمال، هتروزیگوت و هموزیگوت موتانت برای هر دو گروه بیمار و کنترل، فرضیههای پژوهش و بررسی ارتباط بین ژنوتیپ افراد با بروز بیماری از طریق آزمون مجذور کای Chi Square(χ2) محاسبه و موردبررسی و تجزیه تحلیل آماری قرار گرفت. خطای کمتر از 05/0 برای سطح معناداری (05/0> P) در نظر گرفته شد.
نتایج به دست آمده با استفاده از نرمافزار SPSS (version 26) مورد تجزیه و تحلیل آماری قرارگرفت. پس از تعیین فراوانی ژنو تیپهای هموزیگوت نرمال، هتروزیگوت و هموزیگوت موتانت برای هر دو گروه بیمار و کنترل، فرضیههای پژوهش و بررسی ارتباط بین ژنوتیپ افراد با بروز بیماری از طریق آزمون مجذور کای Chi Square(χ2) محاسبه و موردبررسی و تجزیه تحلیل آماری قرار گرفت. خطای کمتر از 05/0 برای سطح معناداری (05/0> P) در نظر گرفته شد.
یافتهها
برای بررسی پلیمورفیسم تک نوکلئوتیدی جایگاه rs1801260 در ژن CLOCK، از تکنیک ARMS-PCR استفاده شد و محصول PCR به طول bp 135 به دست آمد. الکتروفورز محصولات به دست آمده در روش ARMS-PCR برای جفت پرایمر مربوط به این پلیمورفیسم نشان داده است که افراد دارای سه گروه ژنوتیپی هموزیگوت نرمال، هتروزیگوت و هموزیگوت موتانت میباشند (شکل 1). در افراد هموزیگوت نرمال با ژنوتیپ AA، فقط در مخلوط واکنش مربوط به پرایمر نرمال محصول PCR بهصورت تک باند مشاهده شده است. در این افراد واکنش PCR با پرایمر موتانت هیچ محصولی ایجاد نکرده است که نشاندهنده عدم وجود آلل موتانت
G در آنها میباشد.
در افراد هتروزیگوت که دارای ژنوتیپ AG هستند، محصول PCR تک باند در هر دو مخلوط واکنش حاوی پرایمر موتانت و پرایمر نرمال مشاهدهگردید. همچنین در افراد هموزیگوت موتانت با ژنوتیپ GG، فقط در مخلوط واکنش مربوط به پرایمر موتانت محصول PCR بهصورت تک باند به دست آمده است. در این افراد واکنش PCR با پرایمر نرمال هیچ محصولی ایجاد نکرده است که نشاندهنده عدم وجود آلل نرمال A میباشد.
G در آنها میباشد.
در افراد هتروزیگوت که دارای ژنوتیپ AG هستند، محصول PCR تک باند در هر دو مخلوط واکنش حاوی پرایمر موتانت و پرایمر نرمال مشاهدهگردید. همچنین در افراد هموزیگوت موتانت با ژنوتیپ GG، فقط در مخلوط واکنش مربوط به پرایمر موتانت محصول PCR بهصورت تک باند به دست آمده است. در این افراد واکنش PCR با پرایمر نرمال هیچ محصولی ایجاد نکرده است که نشاندهنده عدم وجود آلل نرمال A میباشد.
.jpg)
شکل (1): الکتروفورز محصول ARMS-PCR برای بررسی پلیمورفیسم جایگاه rs1801260 ژن CLOCK. فلش سبز نشاندهنده فرد هموزیگوت سالم AA است، فلش سفید مربوط به فرد هتروزیگوت AG است و فلش زرد رنگ نشاندهنده فرد هموزیگوت بیمار GG است (اندازه باند bp 135، سایز مارکر bp 100).
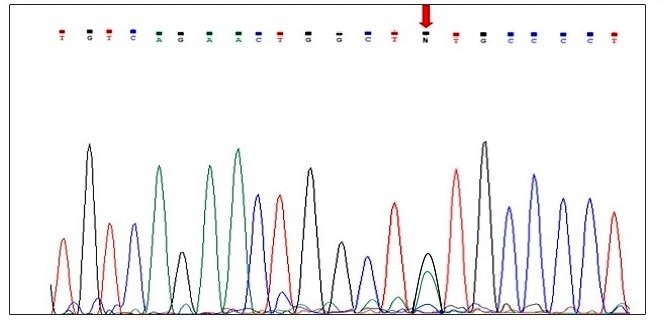
همانطوریکه اشاره شد، برای تأیید نتایج به دست آمده از ARMS-PCR و اطمینان از وجود پلیمورفیسم در جایگاه موردنظر، تعیین توالی تعدادی از محصولات PCR انجام شد. نتایج تعیین توالی محصولات PCR نشان داده است که فرد هوموزیگوت موتانت در جایگاه rs1801260 دارای یک پیک نوکلئوتیدی مربوط به آلل G میباشد و فرد هتروزیگوت دارای دو پیک نوکلئوتیدی است که نشاندهنده وجود هر دو آلل A و G میباشد (شکل 2 و شکل 3).
.jpg)
شکل (2): نتایج تعیین توالی جایگاه rs1801260 در ژن CLOCK. در جایگاه مورد نظر که با فلش نشان داده شده، یک پیک سیاه مربوط به نوکلئوتید G مشاهده میشود که نشاندهنده ژنوتیپ هموزیگوت موتانت با ژنوتیپ GG است.

شکل (3): نتایج تعیین توالی جایگاه rs1801260 در ژن CLOCK. در جایگاه مورد نظر که با فلش نشان داده شده، دو پیک سبز و سیاه مربوط به نوکلئوتیدهای A و G مشاهده میشود که نشاندهنده ژنوتیپ هتروزیگوت AG است.
در این مطالعه 120 نمونه خون (60 بیمار و 60 کنترل نرمال) موردبررسی قرارگرفت. فراوانی و درصد پلیمورفیسم rs1801260 در ژن CLOCK در دو گروه بیمار و کنترل در جدول 2 نشان داده شده است. میزان بروز ژنوتیپ هموزیگوت موتانت GG در افراد بیمار 50درصد و در افراد سالم 30درصد است. میزان بروز ژنوتیپ هتروزیگوت AG در افراد بیمار 7/36درصد و در افراد سالم 7/26درصد است. درحالیکه میزان بروز ژنوتیپ هموزیگوت نرمال AA در افراد بیمار تنها 3/13درصد بوده که این میزان در افراد نرمال به 3/43درصد میرسد (جدول 2)(شکل 4).
جدول (2): فراوانی و درصد CLOCK (rs1801260) در دو گروه بیمار و کنترل CLOCK (rs1801260)
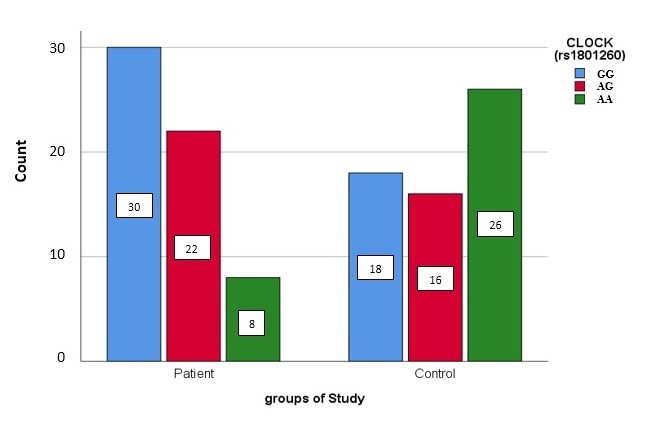 شکل (4): فراوانی ژنوتیپهای هموزیگوت نرمال (AA)، هتروزیگوت (AG) و هموزیگوت موتانت (GG) در جایگاه rs1801260 ژن CLOCK در دو گروه بیمار و کنترل.
شکل (4): فراوانی ژنوتیپهای هموزیگوت نرمال (AA)، هتروزیگوت (AG) و هموزیگوت موتانت (GG) در جایگاه rs1801260 ژن CLOCK در دو گروه بیمار و کنترل.
| ژنوتیپها | بیمار | کنترل | کل | |
| ژنوتیپ GG | تعداد % درصد |
30 % 50 |
18 % 30 |
48 % 40 |
| ژنوتیپ AG | تعداد % درصد |
22 % 36/7 |
16 % 26/7 |
38 % 31/7 |
| ژنوتیپ AA | تعداد % درصد |
8 % 13/3 |
26 % 43/3 |
34 % 28/3 |
| کل | تعداد % درصد |
60 % 100 |
60 % 100 |
120 % 100 |

همانطور که در جدول 3 نشان داده شده است، میزان P value محاسبهشده در آنالیز آماری کمتر از 0/05 به دست آمده است که نشان میدهد بین پلیمورفیسم rs1801260 و خطر بروز بیماری ارتباط معنیداری وجود دارد (0/034 P=). بهعبارتدیگر پلیمورفیسم جایگاه rs1801260 در ژن CLOCK بهطور معنیداری با خطر بروز بیماری مرتبط است.
جدول (3): بررسی رابطه بین پلیمورفیسم rs1801260 و خطر بروز بیماری در دو گروه بیمار و کنترل
| تعداد (درصد) ژنوتیپ rs1801260 |
P-Value |
χ2 |
|||||
| AA | AG | GG | |||||
| نرمال بیمار |
(30%) 18 (50%) 30 |
(267/7%) 16 (36/7%) 22 |
(43/3%) 26 (13/3%) 8 |
6/738 | 0/034 | ||
ارتباط آماری بین جنسیت بیماران و پلیمورفیسم rs1801260 نیز موردبررسی قرار گرفت. همانطور که در جدول 4 نشان داده شده است، ازآنجاکه سطح معنادار آماری (P value) بیشتر از 0/05 در نظر گرفتهشده، بنابراین بین پلیمورفیسم rs1801260 و جنسیت بیماران ارتباط معنیداری وجود ندارد (P=0.924). بهعبارتدیگر پلیمورفیسم rs1801260 بیماران مستقل از جنسیت است. همچنین ارتباط بین سبک زندگی افراد بیماران و وجود پلیمورفیسم rs1801260 ژن CLOCK نیز موردبررسی قرار گرفته است. نتایج حاصله نشان داده است که بین پلیمورفیسم rs1801260 و سبک زندگی بیماران ارتباط معنیداری وجود ندارد (P=0.270). بهعبارتدیگر پلیمورفیسم rs1801260 در بیماران مستقل از سبک زندگی بیماران است (جدول 4). بررسی آماری ارتباط بین سن افراد موردمطالعه و پلیمورفیسم rs1801260 ژن CLOCK نیز نشان داده است که پلیمورفیسم rs1801260 ارتباط معنیداری با سن بیماران ندارد (P=0.943).
جدول (4): بررسی رابطه بین پلیمورفیسم rs1801260 با جنسیت و سبک زندگی بیماران
| تعداد (درصد) ژنوتیپ rs1801260 |
P-Value |
χ2 |
||||
| کل | AA | AG | GG | |||
| مرد زن |
(53/3 %) 16 (46/7 %) 14 |
(33/3 %) 10 (40 %) 12 |
(13/3 %) 4 (13/3 %) 4 |
(100%) 30 (100%) 30 |
0/158 | 0/924 |
| مصرف کافئین بیش از mg300 در روز |
(62/5 %) 20 | (18/8 %) 6 | (18/8 %) 6 | (100 %) 32 | 171/5 | 270/0 |
| مصرف الکل | (40 %) 4 | (60 %) 6 | (0 %) 0 | (100 %)10 | ||
| مشاغل مرتبط با شیفت شب |
(33/3 %) 6 | (55/6 %) 10 | (11/1 %) 2 | (100 %)18 | ||
اختلالات خواب شرایطی هستند که بهواسطه ناهماهنگی بین ریتمهای شبانهروزی داخلی با محیط خارجی به وجود میآیند و اختلال بیخوابی (Insomnia) یکی از مهمترین انواع آن میباشد. اگرچه اثرات محیطی، اجتماعی و یا شغلی ممکن است منجر به اختلال خواب شوند، برخی از افراد نیز ازنظر ژنتیکی مستعد ابتلا به این اختلالات هستند. تخمین زده میشود که حدود 33 درصد از تنوع کیفیت خواب و 40 درصد از تنوع الگوی خواب به علت تفاوتهای ژنتیکی میباشد (1، 5). تنظیم سیستم ساعت شبانهروزی توسط مجموعهای از ژنهای شبانهروزی انجام میشود که ژن CLOCK یکی از مهمترین ژنهای این سیستم بوده و عملکرد اصلی آن فعال سازی رونویسی ژنهای ساعت پایین دست میباشد (5، 6). محصول این ژن یک پروتئین حاوی 846 اسید آمینه است که هم بهعنوان یک فاکتور رونویسی و هم بهعنوان یک استیل ترانسفراز عمل مینماید (1، 7، 19). مطالعات متعدد ارتباط بین پلیمورفیسمهای ژن ساعت شبانهروزی را با ویژگیهای خواب و بیخوابی نشان دادهاند. پلیمورفیسمهای تک نوکلئوتیدی عامل اصلی ایجاد تنوع ژنتیکی در جمعیتها بوده و در ایجاد صفات فنوتیپی خاص موثرند (20). بر اساس نتایج پروژههای ژنوم، در ژن CLOCK تعداد 406 پلیمورفیسم تک نوکلئوتیدی (SNP) در جمعیت آفریقایی، 271 پلیمورفیسم در جمعیت اروپایی، 278 پلیمورفیسم در جمعیت آسیایی و 277 پلیمورفیسم در جمعیت قاره آمریکا شناسایی شده است (21).
لذا در این مطالعه ارتباط بین پلیمورفیسم جایگاه rs1801260 ژن CLOCK با اختلال بیخوابی در جمعیت ایرانی موردبررسی قرار گرفت. بر اساس نتایج این مطالعه، در این جایگاه وجود آلل طبیعی A و آلل موتانت G و نیز ژنوتیپهای هموزیگوت طبیعی (AA)، هتروزیگوت (AG) و هموزیگوت موتانت (GG) در جمعیت مورد مطالعه شناسایی شده است. همچنین نشان داده شد که پلیمورفیسم این جایگاه رابطه معنیداری با اختلال کم خوابی دارد. بطوریکه درصد بیشتری از بیماران دارای ژنوتیپ GG و AG میباشند و به نظر میرسد که وجود ژنوتیپهای مختلف در جایگاه rs1801260 نقش مؤثری در ابتلا به بیماری بیخوابی دارد.
در رابطه با نقش پلیمورفیسم ژنهای تنظیم کننده ساعت شبانهروزی با اختلالات خواب، مطالعه انجام شده توسط Bragantini و همکاران نشان داد که 16 پلیمورفیسم تک نوکلئوتیدی در ژنهای مختلف ساعت ازجمله ژن CRY1 و CLOCK بهطور قابلتوجهی با حداقل 1 علامت بیخوابی مرتبط هستند (22). همچنین سه مطالعه در مقیاس بزرگ از نوع مطالعه گسترده ارتباط ژنومی در جمعیت اروپایی و بریتانیایی انجام شد که در همه موارد سه نوع واریانت ژنتیکی در ژن PER2 بهعنوان فاکتور خطر مرتبط با بیخوابی شناسایی شد (23).
در همین رابطه و هم راستا با نتایج این مطالعه، پلیمورفیسم rs1801260 A/G واقع در ناحیه ترجمه نشدنی (3’-UTR) ژن CLOCK، که منطقه مهمی برای پایداری، بیان و عملکرد mRNA است، بهعنوان اولین پلیمورفیسم مرتبط با اختلالات خواب شناسایی گردید و مشخص شد که در جمعیت اروپایی افراد حامل آلل G در این جایگاه، 10 تا 44 دقیقه تأخیر در زمانبندی ترجیحی برای فاز فعال خواب نشان میدهند (24). با این حال، در برخی مطالعات نیز ارتباط بین این پلیمورفیسم و آلل G با اختلالات خواب و ترجیح خواب عصرگاهی مشاهده نشد که میتواند به دلیل تفاوتها و تنوع ژنتیکی در میان اقوام و جمعیتهای مختلف و یا تأثیر سایر پلیمورفیسمها باشد (25، 26).
در این راستا Mishima و همکاران گزارش دادند که پلیمورفیسم 3111T/C (rs1801260) ژن CLOCK با ترجیح خواب عصر و زمان تأخیر خواب شبانه در جمعیت ژاپنی مرتبط است. بطوریکه هموزیگوت های G/G در مقایسه با افراد حامل آلل A بهطور قابلتوجهی مدت خواب کوتاهتر و افزایش خواب آلودگی در طول روز را نشان میدهند (27). بااینوجود در مطالعهای جدیدتر که توسط Sakurada و همکاران در جمعیت بزرگی (1397 نفر) از بزرگسالان بالای 40 سال در ژاپن انجام شد، ارتباطی بین این پلیمورفیسم با مشکل شروع خواب مشاهده نشد (23).
همچنین در مطالعه انجام شده توسط Garaulet و همکاران نیز نشان داده شد که پلیمورفیسم این جایگاه با اختلال خواب و کم خوابی در جمعیت اسپانیایی مرتبط است (28). درحالیکه مطالعه انجام شده توسط Chang و همکاران و نیز مطالعه Choub و همکاران در جمعیت ایتالیایی و مطالعه pedrazolli و همکاران در جمعیت برزیلی ارتباطی بین پلیمورفیسم rs1801260 و زمان خواب معمولی، فاز شبانهروزی، و یا ریتم شبانهروزی را نشان نداده است (29-31).
همچنین Ning و همکاران با مطالعه روی کارگران چینی شاغل در صنعت نفت نشان دادند که پلیمورفیسم جایگاه rs1801260 ژن CLOCK با ایجاد اختلال خواب در این افراد مرتبط است (32). در مطالعهای دیگر که توسط Shi و همکاران انجام شد، مشخص شد که پلیمورفیسم جایگاه rs1801260 ژن CLOCK بهطور مؤثری با اختلالات خواب و نیز سلامت روان در جمعیت پرستاران چینی ارتباط دارد (33). همچنین مطالعه بر روی جمعیت زنان یائسه قفقازی و آسیایی مبتلا به بیخوابی نیز ارتباط بین این پلیمورفیسم را با ریتم شبانهروزی ملاتونین نشان داده است (34).
از سوی دیگر مطالعهای بر روی موشها نشان داد که پلیمورفیسم جایگاه rs1801260 ژن CLOCK منجر به تفاوت در بیان این ژن بر اساس ژنوتیپ میشود، بهطوری که در موشهای حامل آلل جهش یافته، بیان بیشتر پروتئین CLOCK مشاهده شد (15). بیان بالاتر CLOCK در حضور آلل G در برخی ردههای سلولی مختلف انسانی نیز مشاهده شده است. از آنجاییکه این پلیمورفیسم تک نوکلئوتیدی در یک جایگاه میانکنش با miRNA-182 قرار دارد میتواند نتایج مربوط به کاهش بیان آن را توجیه نماید (35). بااینوجود در مطالعهای که توسط Barragan و همکاران در سال 2022 بر روی جمعیت افراد سالم اسپانیایی انجام شد مشخص شد که پلیمورفیسم rs1801260 با میزان بیان ژن CLOCK ارتباط مؤثری ندارد (14).
برخی مطالعات نیز ارتباط پلیمورفیسم rs1801260 را با سایر اختلالات و بیماریها ازجمله اختلال بیش فعالی نشان دادهاند (36 و 37). مطالعه انجامشده توسط Pagliai و همکاران بر روی جمعیت ایتالیایی نشان داده است که افراد هموزیگوت برای آلل جهشیافته (GG) در جایگاه rs1801260 کمتر اضافهوزن دارند و دارای سطوح گلوکز ناشتا بالاتر و میزان افسردگی سالمندی بالاتری هستند (12). همچنین شواهد متعدد حاصل از مطالعات مولکولی و مدلهای حیوانی نشان دادهاند که اختلال در سیکل شبانهروزی و ژنهای مسئول آن با بیماریهای تحلیل برنده سیستم عصبی همچون پارکینسون نیز مرتبط هستند (38 و 39). برخی مطالعات نیز ارتباط بین سایر پلیمورفیسمهای ژن CLOCK شامل rs2070062 و rs6853192 و rs6820823 و rs3792603 و rs11726609 را با اختلالات خواب و افزایش یا کاهش مدتزمان خواب نشان دادهاند (17، 40).
تنها گزارش مربوط به ایران نیز ارتباط پلیمورفیسم rs1801260 3111 A/G ژن CLOCK را با میزان هورمون گرسنگی، زمانبندی غذا خوردن و میزان و الگوی خواب در بزرگسالان ایرانی دارای اضافهوزن و چاق بررسی نموده و نشان داده است که پلیمورفیسم این جایگاه با بسیاری از پارامترهای رفتاری این افراد و ازجمله کاهش میزان خواب و الگوی خواب افراد رابطه معنیداری دارد (41).
درمجموع با توجه به اهمیت ژن CLOCK بهعنوان یکی از مهمترین ژنهای ساعت در انسان که در توازن و زمانبندی ریتمهای بیولوژیکی در سطح مولکولی نقش دارد، به نظر میرسد که هرگونه تغییر در این ژن شامل وقوع جهشها و یا وجود پلیمورفیسمها میتواند باعث بروز اختلال بیخوابی شود. نتایج بهدستآمده در این مطالعه نیز نشان داده است که پلیمورفیسم جایگاه rs1801260 ژن CLOCK ارتباط معنیدار و مؤثری با بروز اختلال بیخوابی در بیماران ایرانی دارد. همچنین بر اساس نتایج پلیمورفیسم این جایگاه با جنسیت، سن، و سبک زندگی بیماران ارتباط معنیداری ندارد. بااینوجود برای دستیابی به نتایج دقیقتر، پیشنهاد میشود که بررسیهای بیشتر در این زمینه بر روی تعداد بیشتری از بیماران و در جمعیت آماری بزرگتر انجام شود. همچنین انجام بررسی در سایر استانها و مناطق جغرافیایی ایران و نیز بررسی سایر پلیمورفیسمهای ژن CLOCK میتواند موردتوجه قرار گیرد.
لذا در این مطالعه ارتباط بین پلیمورفیسم جایگاه rs1801260 ژن CLOCK با اختلال بیخوابی در جمعیت ایرانی موردبررسی قرار گرفت. بر اساس نتایج این مطالعه، در این جایگاه وجود آلل طبیعی A و آلل موتانت G و نیز ژنوتیپهای هموزیگوت طبیعی (AA)، هتروزیگوت (AG) و هموزیگوت موتانت (GG) در جمعیت مورد مطالعه شناسایی شده است. همچنین نشان داده شد که پلیمورفیسم این جایگاه رابطه معنیداری با اختلال کم خوابی دارد. بطوریکه درصد بیشتری از بیماران دارای ژنوتیپ GG و AG میباشند و به نظر میرسد که وجود ژنوتیپهای مختلف در جایگاه rs1801260 نقش مؤثری در ابتلا به بیماری بیخوابی دارد.
در رابطه با نقش پلیمورفیسم ژنهای تنظیم کننده ساعت شبانهروزی با اختلالات خواب، مطالعه انجام شده توسط Bragantini و همکاران نشان داد که 16 پلیمورفیسم تک نوکلئوتیدی در ژنهای مختلف ساعت ازجمله ژن CRY1 و CLOCK بهطور قابلتوجهی با حداقل 1 علامت بیخوابی مرتبط هستند (22). همچنین سه مطالعه در مقیاس بزرگ از نوع مطالعه گسترده ارتباط ژنومی در جمعیت اروپایی و بریتانیایی انجام شد که در همه موارد سه نوع واریانت ژنتیکی در ژن PER2 بهعنوان فاکتور خطر مرتبط با بیخوابی شناسایی شد (23).
در همین رابطه و هم راستا با نتایج این مطالعه، پلیمورفیسم rs1801260 A/G واقع در ناحیه ترجمه نشدنی (3’-UTR) ژن CLOCK، که منطقه مهمی برای پایداری، بیان و عملکرد mRNA است، بهعنوان اولین پلیمورفیسم مرتبط با اختلالات خواب شناسایی گردید و مشخص شد که در جمعیت اروپایی افراد حامل آلل G در این جایگاه، 10 تا 44 دقیقه تأخیر در زمانبندی ترجیحی برای فاز فعال خواب نشان میدهند (24). با این حال، در برخی مطالعات نیز ارتباط بین این پلیمورفیسم و آلل G با اختلالات خواب و ترجیح خواب عصرگاهی مشاهده نشد که میتواند به دلیل تفاوتها و تنوع ژنتیکی در میان اقوام و جمعیتهای مختلف و یا تأثیر سایر پلیمورفیسمها باشد (25، 26).
در این راستا Mishima و همکاران گزارش دادند که پلیمورفیسم 3111T/C (rs1801260) ژن CLOCK با ترجیح خواب عصر و زمان تأخیر خواب شبانه در جمعیت ژاپنی مرتبط است. بطوریکه هموزیگوت های G/G در مقایسه با افراد حامل آلل A بهطور قابلتوجهی مدت خواب کوتاهتر و افزایش خواب آلودگی در طول روز را نشان میدهند (27). بااینوجود در مطالعهای جدیدتر که توسط Sakurada و همکاران در جمعیت بزرگی (1397 نفر) از بزرگسالان بالای 40 سال در ژاپن انجام شد، ارتباطی بین این پلیمورفیسم با مشکل شروع خواب مشاهده نشد (23).
همچنین در مطالعه انجام شده توسط Garaulet و همکاران نیز نشان داده شد که پلیمورفیسم این جایگاه با اختلال خواب و کم خوابی در جمعیت اسپانیایی مرتبط است (28). درحالیکه مطالعه انجام شده توسط Chang و همکاران و نیز مطالعه Choub و همکاران در جمعیت ایتالیایی و مطالعه pedrazolli و همکاران در جمعیت برزیلی ارتباطی بین پلیمورفیسم rs1801260 و زمان خواب معمولی، فاز شبانهروزی، و یا ریتم شبانهروزی را نشان نداده است (29-31).
همچنین Ning و همکاران با مطالعه روی کارگران چینی شاغل در صنعت نفت نشان دادند که پلیمورفیسم جایگاه rs1801260 ژن CLOCK با ایجاد اختلال خواب در این افراد مرتبط است (32). در مطالعهای دیگر که توسط Shi و همکاران انجام شد، مشخص شد که پلیمورفیسم جایگاه rs1801260 ژن CLOCK بهطور مؤثری با اختلالات خواب و نیز سلامت روان در جمعیت پرستاران چینی ارتباط دارد (33). همچنین مطالعه بر روی جمعیت زنان یائسه قفقازی و آسیایی مبتلا به بیخوابی نیز ارتباط بین این پلیمورفیسم را با ریتم شبانهروزی ملاتونین نشان داده است (34).
از سوی دیگر مطالعهای بر روی موشها نشان داد که پلیمورفیسم جایگاه rs1801260 ژن CLOCK منجر به تفاوت در بیان این ژن بر اساس ژنوتیپ میشود، بهطوری که در موشهای حامل آلل جهش یافته، بیان بیشتر پروتئین CLOCK مشاهده شد (15). بیان بالاتر CLOCK در حضور آلل G در برخی ردههای سلولی مختلف انسانی نیز مشاهده شده است. از آنجاییکه این پلیمورفیسم تک نوکلئوتیدی در یک جایگاه میانکنش با miRNA-182 قرار دارد میتواند نتایج مربوط به کاهش بیان آن را توجیه نماید (35). بااینوجود در مطالعهای که توسط Barragan و همکاران در سال 2022 بر روی جمعیت افراد سالم اسپانیایی انجام شد مشخص شد که پلیمورفیسم rs1801260 با میزان بیان ژن CLOCK ارتباط مؤثری ندارد (14).
برخی مطالعات نیز ارتباط پلیمورفیسم rs1801260 را با سایر اختلالات و بیماریها ازجمله اختلال بیش فعالی نشان دادهاند (36 و 37). مطالعه انجامشده توسط Pagliai و همکاران بر روی جمعیت ایتالیایی نشان داده است که افراد هموزیگوت برای آلل جهشیافته (GG) در جایگاه rs1801260 کمتر اضافهوزن دارند و دارای سطوح گلوکز ناشتا بالاتر و میزان افسردگی سالمندی بالاتری هستند (12). همچنین شواهد متعدد حاصل از مطالعات مولکولی و مدلهای حیوانی نشان دادهاند که اختلال در سیکل شبانهروزی و ژنهای مسئول آن با بیماریهای تحلیل برنده سیستم عصبی همچون پارکینسون نیز مرتبط هستند (38 و 39). برخی مطالعات نیز ارتباط بین سایر پلیمورفیسمهای ژن CLOCK شامل rs2070062 و rs6853192 و rs6820823 و rs3792603 و rs11726609 را با اختلالات خواب و افزایش یا کاهش مدتزمان خواب نشان دادهاند (17، 40).
تنها گزارش مربوط به ایران نیز ارتباط پلیمورفیسم rs1801260 3111 A/G ژن CLOCK را با میزان هورمون گرسنگی، زمانبندی غذا خوردن و میزان و الگوی خواب در بزرگسالان ایرانی دارای اضافهوزن و چاق بررسی نموده و نشان داده است که پلیمورفیسم این جایگاه با بسیاری از پارامترهای رفتاری این افراد و ازجمله کاهش میزان خواب و الگوی خواب افراد رابطه معنیداری دارد (41).
درمجموع با توجه به اهمیت ژن CLOCK بهعنوان یکی از مهمترین ژنهای ساعت در انسان که در توازن و زمانبندی ریتمهای بیولوژیکی در سطح مولکولی نقش دارد، به نظر میرسد که هرگونه تغییر در این ژن شامل وقوع جهشها و یا وجود پلیمورفیسمها میتواند باعث بروز اختلال بیخوابی شود. نتایج بهدستآمده در این مطالعه نیز نشان داده است که پلیمورفیسم جایگاه rs1801260 ژن CLOCK ارتباط معنیدار و مؤثری با بروز اختلال بیخوابی در بیماران ایرانی دارد. همچنین بر اساس نتایج پلیمورفیسم این جایگاه با جنسیت، سن، و سبک زندگی بیماران ارتباط معنیداری ندارد. بااینوجود برای دستیابی به نتایج دقیقتر، پیشنهاد میشود که بررسیهای بیشتر در این زمینه بر روی تعداد بیشتری از بیماران و در جمعیت آماری بزرگتر انجام شود. همچنین انجام بررسی در سایر استانها و مناطق جغرافیایی ایران و نیز بررسی سایر پلیمورفیسمهای ژن CLOCK میتواند موردتوجه قرار گیرد.
نوع مطالعه: پژوهشي(توصیفی- تحلیلی) |
موضوع مقاله:
ژنتیک
فهرست منابع
1. Liu C, Tang X, Gong Z, Zeng W, Hou Q, Lu R. Circadian Rhythm Sleep Disorders: Genetics, Mechanisms, and Adverse Effects on Health. Front Genet 2022;13:875342. [DOI:10.3389/fgene.2022.875342] [PMID] [PMCID]
2. Meyer N, Harvey AG, Lockley SW, Dijk DJ. Circadian rhythms and disorders of the timing of sleep. Lancet 2022;400(10357):1061-78.
https://doi.org/10.1016/S0140-6736(22)00877-7 [DOI:10.1016/s0140-6736(22)00877-7.] [PMID]
3. Morin CM, Jarrin DC. Epidemiology of Insomnia: Prevalence, Course, Risk Factors, and Public Health Burden. Sleep Med Clin 2022;17(2):173-91. [DOI:10.1016/j.jsmc.2022.03.003] [PMID]
4. Kapoor V, Watson NF, Ball L. Chronic insomnia in the setting of MTHFR polymorphism. J Clin Sleep Med 2022;18(4):1215-18. [DOI:10.5664/jcsm.9794] [PMID] [PMCID]
5. Ashbrook LH, Krystal AD, Fu YH, Ptáček LJ. Genetics of the human circadian clock and sleep homeostat. Neuropsychopharmacology 2020;45(1):45-54. [DOI:10.1038/s41386-019-0476-7] [PMID] [PMCID]
6. Patke A, Young MW, Axelrod S. Molecular mechanisms and physiological importance of circadian rhythms. Nat Rev Mol Cell Biol 2020;21(2):67-84. [DOI:10.1038/s41580-019-0179-2] [PMID]
7. Takahashi JS. Transcriptional architecture of the mammalian circadian clock. Nat Rev Genet 2017;18(3):164-79. [DOI:10.1038/nrg.2016.150] [PMID] [PMCID]
8. Lateef OM, Akintubosun MO. Sleep and Reproductive Health. J Circadian Rhythms 2020;18:1. [DOI:10.5334/jcr.190] [PMID] [PMCID]
9. Rijo-Ferreira F, Takahashi JS. Genomics of Circadian Rhythms in Health and Disease. Genome Med 2009;11:82. [DOI:10.1186/s13073-019-0704-0] [PMID] [PMCID]
10. Möller-Levet CS, Archer SN, Bucca G, Laing EE, Slak A, Kabiljo R, et al. Effects of insufficient sleep on circadian rhythmicity and expression amplitude of the human blood transcriptome. Proc Natl Acad Sci USA 2013;110(12):e1132-41. [DOI:10.1073/pnas.1217154110] [PMID] [PMCID]
11. Carroll JE, Cole SW, Seeman TE, Breen EC, Witarama T, Arevalo JMG, et al. Partial sleep deprivation activates the DNA damage response (DDR) and the senescence-associated secretory phenotype (SASP) in aged adult humans. Brain Behav Immun 2016;2(51):223-9. [DOI:10.1016/j.bbi.2015.08.024] [PMID] [PMCID]
12. Pagliai G, Sofi F, Dinu M, Sticchi E, Vannetti F, Molino Lova R, et al. CLOCK gene polymorphisms and quality of aging in a cohort of nonagenarians - The MUGELLO Study. Sci Rep 2019;9(1):1472. [DOI:10.1038/s41598-018-37992-8] [PMID] [PMCID]
13. Chai R, Liao M, Ou L, Tang Q, Liang Y, Li N, et al. Circadian Clock Genes Act as Diagnostic and Prognostic Biomarkers of Glioma: Clinic Implications for Chronotherapy. Biomed Res Int 2022;2022:9774879. [DOI:10.1155/2022/9774879] [PMID] [PMCID]
14. Barragán R, Sorlí JV, Coltell O, Gonzalez-Monje I, Fernández-Carrión R, Villamil LV, et al. Influence of DNA-Polymorphisms in Selected Circadian Clock Genes on Clock Gene Expression in Subjects from the General Population and Their Association with Sleep Duration. Medicina (Kaunas) 2022;16;58(9):1294. [DOI:10.3390/medicina58091294] [PMID] [PMCID]
15. Ozburn AR, Purohit K, Parekh PK., Kaplan GN, Falcon E, Mukherjee S, et al. Functional implications of the CLOCK 3111T/C single-nucleotide polymorphism. Front Psychiatry 2016;7:67. [DOI:10.3389/fpsyt.2016.00067] [PMID] [PMCID]
16. Kripke DF, Nievergelt CM, Joo, E, Shekhtman T, Kelsoe JR. (2009). Circadian polymorphisms associated with affective disorders. J Circadian Rhythms 2009;7(2);3-25. [DOI:10.1186/1740-3391-7-2] [PMID] [PMCID]
17. Zhang L, Ptáček LJ, Fu YH. Diversity of human clock genotypes and consequences. Prog Mol Biol Transl Sci 2013;119:51-81. [DOI:10.1016/B978-0-12-396971-2.00003-8] [PMID] [PMCID]
18. Naylor E, Bergmann BM, Krauski K, et al. The circadian clock mutation alters sleep homeostasis in the mouse. J Neurosci 2000;20(21):8138-43.
https://doi.org/10.1523/JNEUROSCI.20-21-08138.2000 [DOI:10.1523/jneurosci.20-21-08138.2000] [PMID] [PMCID]
19. Doi M, Hirayama J, Sassone-Corsi P. Circadian Regulator Clock Is a Histone Acetyltransferase. Cell 2006;125(3):497-508. [DOI:10.1016/j.cell.2006.03.033] [PMID]
20. Fedorova L, Khrunin A, Khvorykh G, Lim J, Thornton N, Mulyar OA, et al. Analysis of Common SNPs across Continents Reveals Major Genomic Differences between Human Populations. Genes (Basel) 2022;13(8):1472. [DOI:10.3390/genes13081472] [PMID] [PMCID]
21. The 1000 Genomes Project Consortium. A global reference for human genetic variation.Nature variation. Nature 2015; 526:68-74. [URL]
22. Bragantini D, Sivertsen B, Gehrman P, Lydersen S, Guzey IC. Variations in circadian genes and individual nocturnal symptoms of insomnia. The HUNT study. Chronobiol Int 2019;36:681-88. [DOI:10.1080/07420528.2019.1582540] [PMID]
23. Sakurada K, Konta T, Takahashi S, Murakami N, Sato H, Murakami R, et al. Circadian Clock Gene Polymorphisms and Sleep-Onset Problems in a Population-Based Cohort Study: The Yamagata Study. Tohoku J Exp Med 2021;255(4):325-31. [DOI:10.1620/tjem.255.325] [PMID]
24. Katzenberg D, Young T, Finn L, et al. A CLOCK polymorphism associated with human diurnal preference. Sleep 1998;21(6):569-76. [DOI:10.1093/sleep/21.6.569] [PMID]
25. Allebrandt KV, Roenneberg T. The search for circadian clock components in humans: new perspectives for association studies. Braz J Med Biol Res 2008;41(8):716-21.
https://doi.org/10.1590/S0100-879X2008000800013 [DOI:10.1590/s0100-879x2008000800013] [PMID]
26. von Schantz M. Phenotypic effects of genetic variability in human clock genes on circadian and sleep parameters. J Genet 2008;87(5):513-19. [DOI:10.1007/s12041-008-0074-7] [PMID]
27. Mishima K, Tozawa T, Satoh K, Saitoh H, Mishima Y. The 3111T/C polymorphism of hClock is associated with evening preference and delayed sleep timing in a Japanese population sample. Am J Med Genet B Neuropsychiatr Genet 2005;133(1):101-4. [DOI:10.1002/ajmg.b.30110] [PMID]
28. Garaulet M, Sanchez-Moreno C, Smith CE, Lee YC, Nicolas F, Ordovas JM. Ghrelin, sleep reduction and evening preference: relationships to CLOCK 3111 T/C SNP and weight loss. PLoS One 2011;6(2):e17435. [DOI:10.1371/journal.pone.0017435] [PMID] [PMCID]
29. Chang AM, Buch AM, Bradstreet DS, Klements DJ, Duffy JF. Human diurnal preference and circadian rhythmicity are not associated with the CLOCK 3111C/T gene polymorphism. J Biol Rhythms 2011;26(3):276-9. [DOI:10.1177/0748730411402026] [PMID] [PMCID]
30. Choub A, Mancuso M, Coppede F, LoGerfo A, Orsucci D, Petrozzi L, et al. Clock T3111C and Per2 C111G SNPs do not influence circadian rhythmicity in healthy Italian population. Neurol Sci 2011;32:89-93. [DOI:10.1007/s10072-010-0415-1] [PMID]
31. Pedrazzoli M, Louzada FM, Pereira DS, Benedito-Silva AA, Lopez AR, Martynhak BJ, et al. Clock polymorphisms and circadian rhythms phenotypes in a sample of the Brazilian population. Chronobiol Int 2007;24(ja 2007):1-8. [DOI:10.1080/07420520601139789] [PMID]
32. Ning L, Shi L, Tao N, Li R, Jiang T, Liu J. Effects of Occupational Stress and Circadian CLOCK Gene Polymorphism on Sleep Quality of Oil Workers in Xinjiang, China. Med Sci Monit 2020;26:e924202.
https://doi.org/10.12659/MSM.924202 [DOI:10.12659/msm.924202]
33. Shi L, Liu Y, Jiang T, Yan P, Cao F, Chen Y, et al. Relationship between Mental Health, the CLOCK Gene, and Sleep Quality in Surgical Nurses: A Cross-Sectional Study. Biomed Res Int 2020;2020:4795763. [DOI:10.1155/2020/4795763] [PMID] [PMCID]
34. Semenova NV, Madaeva IM, Bairova TA, Zhambalova RM, Sholokhov LF, Kolesnikova LI. Association of the melatonin circadian rhythms with clock 3111T/C gene polymorphism in Caucasian and Asian menopausal women with insomnia. Chronobiol Int 2018;35:1066-1076. [DOI:10.1080/07420528.2018.1456447] [PMID]
35. Saus E, Soria V, Escaramis G, Vivarelli F, Crespo JM, Kagerbauer B, et al. Genetic variants and abnormal processing of pre-miR-182, a circadian clock modulator. In major depression patients with late insomnia. Hum Mol Genet 2010;19,4017-25. [DOI:10.1093/hmg/ddq316] [PMID]
36. Xu X, Breen G, Chen CK, Huang YS, Wu YY, Asherson P. Association study between a polymorphism at the 3'-untranslated region of CLOCK gene and attention deficit hyperactivity disorder. Behav Brain Funct 2010;6:48. [DOI:10.1186/1744-9081-6-48] [PMID] [PMCID]
37. Carpena MX, Hutz MH, Salatino-Oliveira A, Polanczyk GV, Zeni C, Schmitz M, et al. CLOCK Polymorphisms in Attention-Deficit/Hyperactivity Disorder (ADHD): Further Evidence Linking Sleep and Circadian Disturbances and ADHD. Genes 2019;10(2):88. [DOI:10.3390/genes10020088] [PMID] [PMCID]
38. Hunt J., Coulson E.J., Rajnarayanan R., Oster H., Videnovic A., Rawashdeh O. Sleep and circadian rhythms in Parkinson's disease and preclinical models. Mol Neurodegener 2022;17:2. [DOI:10.1186/s13024-021-00504-w] [PMID] [PMCID]
39. Leng Y., Musiek E.S., Hu K., Cappuccio F.P., Yaffe K. Association between circadian rhythms and neurodegenerative diseases. Lancet Neurol. 2019;18:307-18.
https://doi.org/10.1016/S1474-4422(18)30461-7 [DOI:10.1016/s1474-4422(18)30461-7] [PMID]
40. Riestra P, Gebreab SY, Xu R, Khan RJ, Gaye A, Correa A, et al. Circadian CLOCK gene polymorphisms in relation to sleep patterns and obesity in African Americans: findings from the Jackson heart study. BMC Genet 2017;18(1):58. [DOI:10.1186/s12863-017-0522-6] [PMID] [PMCID]
41. Rahati S, Qorbani M, Naghavi A, Heidari nia M, Pishvahm, H. Association between CLOCK 3111 T/C polymorphism with ghrelin, GLP-1, food timing, sleep and chronotype in overweight and obese Iranian adults. BMC Endocr Disord 2022; 22. [DOI:10.1186/s12902-022-01063-x] [PMID] [PMCID]
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |







